@lakesea
2018-12-05T03:17:44.000000Z
字数 1169
阅读 746
Week43:世界范围的油菜种的全基因组重新测序揭示了其生态型分歧的遗传基础
文献解读
今天给大家介绍一篇,最近发布的与群体遗传相关的比较大型的油菜重测序文章。
主要内容
在这项研究中,研究者重新测序并发现了991种天然油菜籽的遗传多样性,其中大部分来自主要的油菜籽生产区域(即欧洲国家,中国,加拿大和澳大利亚)。将这些材料的reads,比对到两个不同的参考基因组(‘Darmor-bzh’ and ‘Tapidor’)中进行群体遗传学的分析,并且其他们发现这通过两组SNP做出来的结果具有高度一致性。
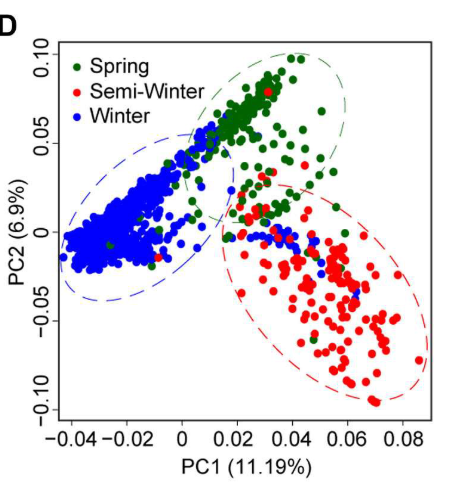
根据PCA的结果,这991个油菜材料可以大概分为三组,相对应了三种生态型,winter, semi-winter and spring。
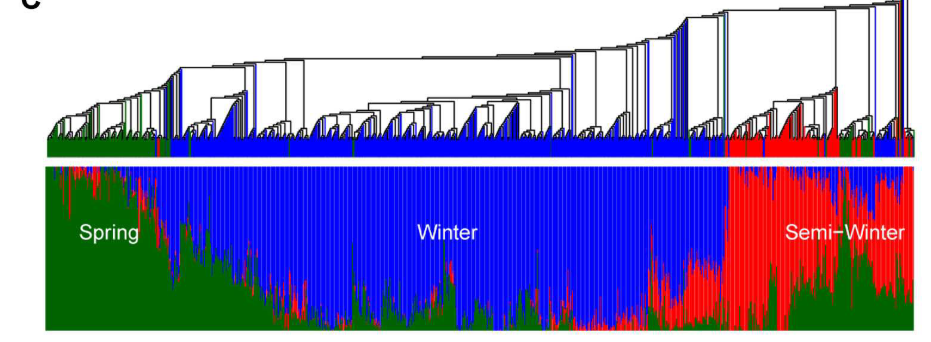
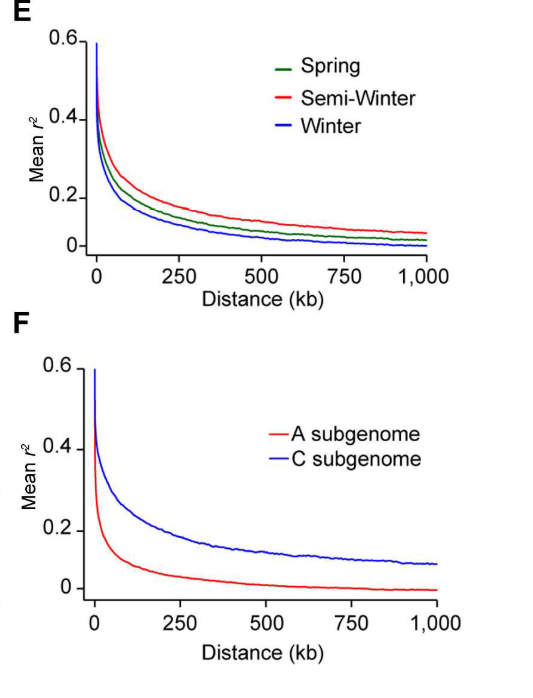
然后研究者发现SNPs和InDels的密度在A subgenome上要比C subgenome更加高,这说明A subgenome的遗传多样性更高。并且在A subgenome中其LD衰减的速度更加快,也说了其重组率更高。为什么会产生这个差别呢,文中提到A 基因组是由受到来自B.rapa引入(更多多样性),而C基因组多样性或多或少地受限于,因为它是源于该物种的原始异源多倍化事件所产生的。
总的来说,油菜的育种还是受限于其比较低的基因多样性,通过人工合成是增加油菜基因多样性的方法之一。
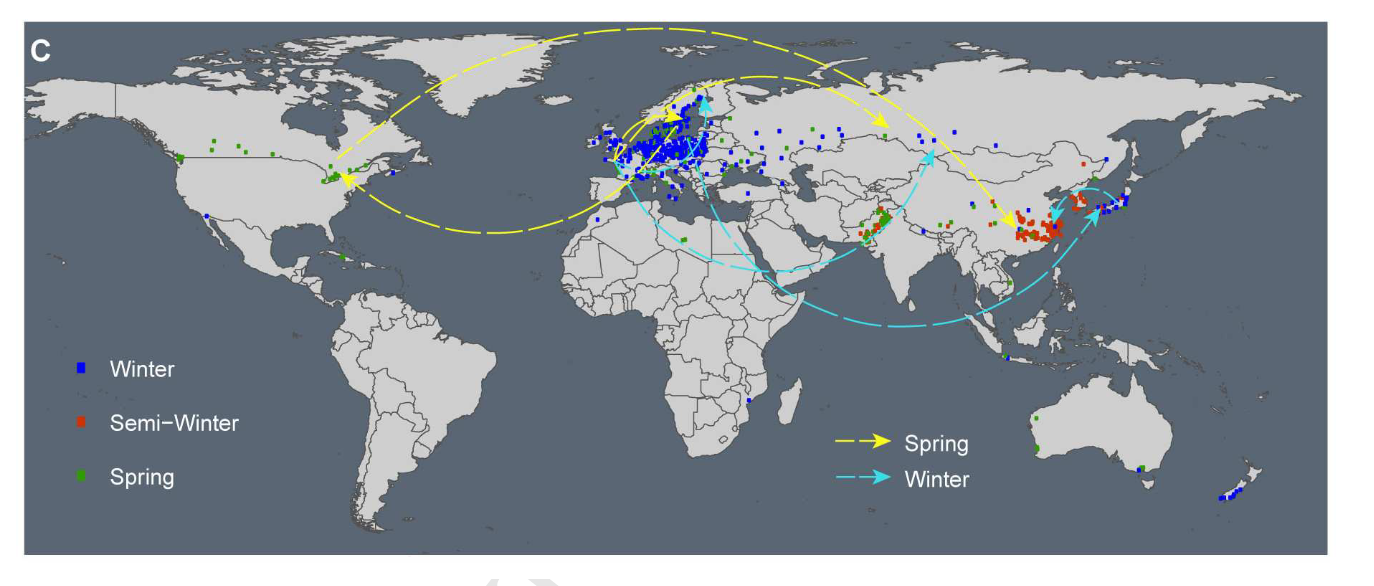
接下来研究者讨论了油菜的遗传漂流。三条比较主要的基因漂流路线被发现:
- winter-type的油菜的基因漂流从法国到俄罗斯,瑞士或者德国等欧洲其他地方。
- 日本到中国的winter-type的油菜基因漂流
- 加拿大到中国的spring-type油菜的基因漂流
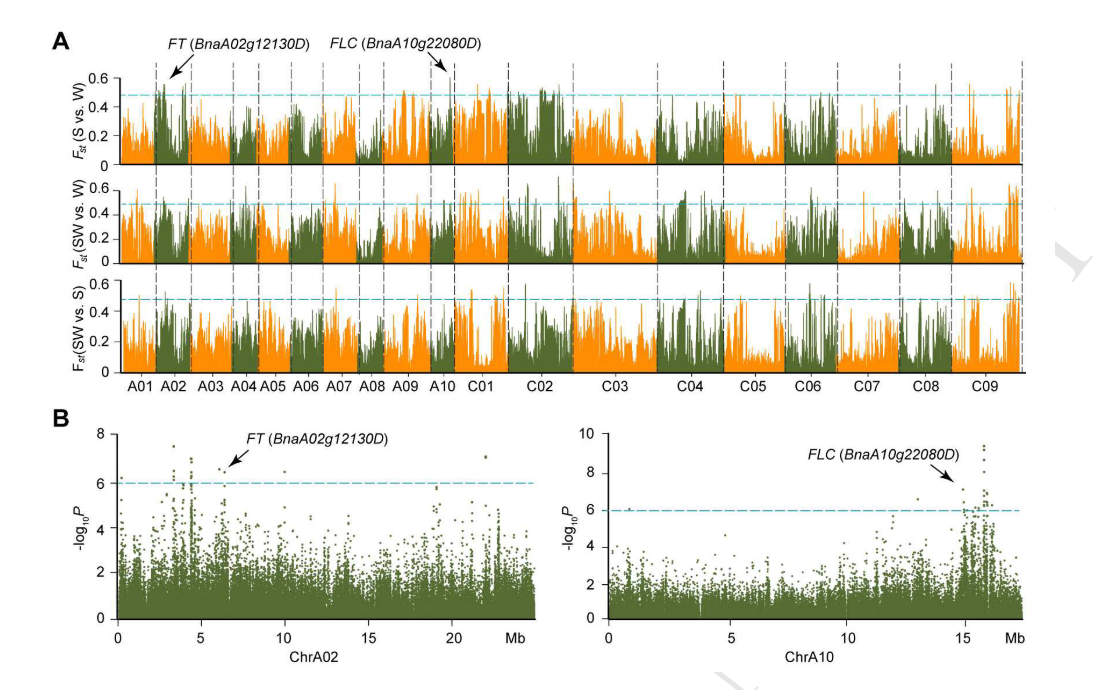
三种不同生态型的油菜是自然选择和人工选择的共同结果.研究者接着进行了选择压力的分析,并发现从winter type到spring type,共有1404个位点 和61选择性清除。这些被选择的基因包括与开花时间相关的基因和与乙烯生物合成或者对应信号通路相关的基因。因为季节的变化,油菜需要缩短其生长周期和开花的时间,部分解析了为什么在这两种生态型间,这些基因会受到选择。
接着研究者再通过GWAS去验证这些受选择的基因是否与开花时间,乙烯生物合作等形状相关。另外研究者还通过转录组研究发现,相对于其他生态型来说,与开花时间相关的FT和FLC基因在spring-type中分别表达高和低。研究者认为,FT和FLC对应的基因表达差异是由,不同生态型中特异性SNP可以促成它们的差异表达,从而控制开花时间以响应三种生态型在地理上适应的各种环境条件。
最后作者对该研究的不足进行了简单的总结,由于测序深度不够,该文章无法进对这三种不同类型的油菜进一步进行基因的PAV和结构变异的研究。
小结:
该文章的值得学习的地方是,进化选择分析做的很充分,然后再进一步讨论是结合已经的信息对受选择的位点和与GWAS的位点进行讨论。