@lakesea
2018-05-02T09:06:56.000000Z
字数 2794
阅读 1790
Week10-水稻泛基因组浏览器(含括3000份水稻)
最近在起稿一篇review paper,主要是关于Pangenome and crop breeding,中心就是如何将泛基因组运用到作物育种中。今天这篇文章,我想就是一个很好的例子,能够将找到的泛基因组很好的展现在公众中,进而方便育种家或者植物学家利用这些新找到的基因。如果大家不知道什么是泛基因组,也欢迎大家阅读我之前写的关于泛基因的文章一文带你了解泛基因组
题目:RPAN: rice pan-genome browser for ∼3000 rice genome
Abstract
在3000多个水稻基因组计划中(3K RGP)研究者测序了3000多个水稻基因组,平均测序深度为14.3×,为水稻研究提供了巨大的资源。在本文中,研究者提供了一个基因组浏览器Rice Pan-genome Browser(RPAN),用于搜索和可视化来自3K RGP的水稻泛基因组。 RPAN包含3010份水稻基因组的基本信息的数据库,包括水稻泛基因组的基因组序列,基因注释,PAV信息和基因表达数据。包括参考基因组中缺少至少12 000个新基因。 RPAN还提供了多种搜索和可视化功能。 浏览器包含3010测序水稻种质,基因组序列,基因注释以及基因表达的信息。它还提供了几种搜索功能,例如PAV信息的搜索功能以及水稻种质或基因ID列表。此外,它还提供了针对水稻泛基因组的特定DNA序列的搜索。RPAN提供的数据资源和工具将加速水稻生物学的基础研究和水稻育种的应用工作。RPAN可以成为水稻生物学和水稻育种的丰富资源。
Methods
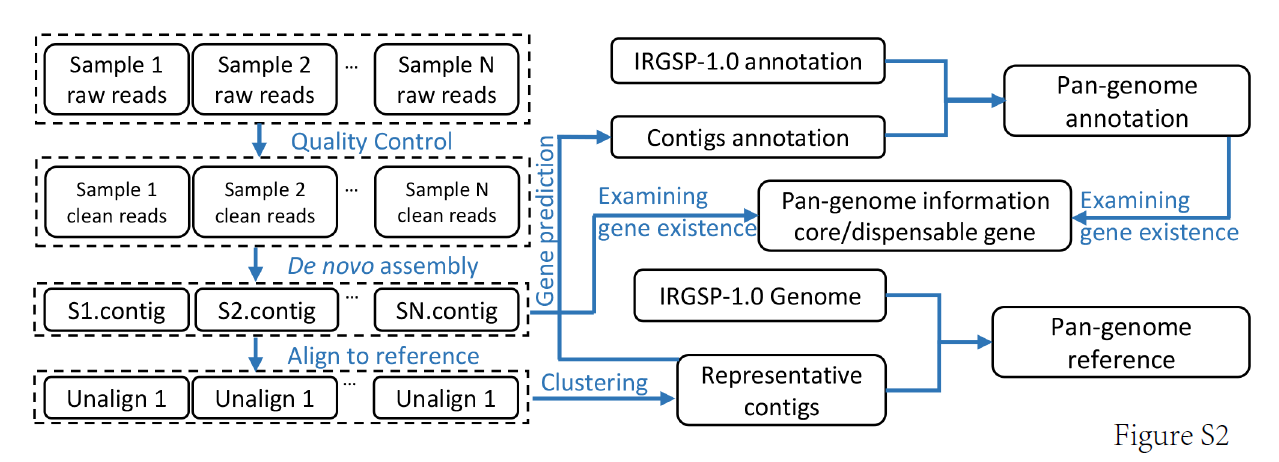
总体构建pangenome的流程如下:
Pan-genome construction
先用soap denovo 分别每一个individual进行assembly(??深度足够吗),然后将assembly 比对到原来的ref中(使用 nucmer tool in Mummer ),找到比对不上的contigs,对这些contigs。然后使用blast去掉有污染的contigs,使用all-blast-all去掉重复的contigs。根据SNP calling的结果,对新的contigs进行分组。
Pan-genome annotation
对那些过滤后的contigs进行基因注释,使用MAKER-P。
PAV determination
基因的深度用来决定这个基因是present or absent。使用简单的bwa mapping,然后samtools 就足够了。编码区覆盖> 0.95且基因区域覆盖> 0.85的基因被认为是present的。
表达量数据
从所有可用的公共数据库,收集了来自不同水稻组织的RNA-seq数据的226个,包括幼芽,孕穗,愈伤组织和四叶阶段枝条。首先用Trimmomatic修剪获得的RNA-seq数据,参数为ILLUMINACLIP:2:30:10 LEADING:20 TRAILING:20 SLIDINGWINDOW:4:20 MINLEN:36',这产生了干净的RNA-seq数据大小为3.4TB。然后将RNA-seq数据与HISAT2与稻泛基因组进行比对。对齐结果被转换,排序并以samtools以.bam文件格式存储。在bedtools中,每个基因的覆盖率计算为'bedtools覆盖率'。
可视化
可视化页面由两部分组成。左侧面板中的树形浏览器和右侧面板中的基因组浏览器。基因组浏览器是基于JBrowse框架构建的,树形浏览器是在HTML5,SVG和JavaScript内部实现的。
Results
用IRGSP-1.0基因组和参考基因组中未包括的所有新序列构建水稻泛基因组。根据源自SNPs的系统发生树,根据其来源水稻种质将这些新序列分组。水稻种质的一个亚群中的所有新序列被连接为一个假染色体。将所有3010份水稻种质的测序数据映射到泛基因组并通过JBrowse框架可视化。
- RPAN数据库还包括大于3000种水稻种质的基本信息,全基因组表达谱数据,基因注释以及存在与否的变异。来自3K RGP的3010个水稻种质的基本信息,包括IRGSP-1.0基因组的登录名,测序深度,图谱深度以及地理位置,亚种(或亚群)分类等元信息。
- 编码序列,蛋白质序列和在水稻泛基因组中的50个995全长编码基因的注释。
- 基因存在 - 缺乏变异(PAV)。通过453个高品质材料确定了水稻泛基因组中基因的存在/缺失。然后将所有基因分类为核心,候选核心或不同类型的分布基因。总共有23 914个核心基因,4986个候选核心基因和22 095个分布基因。在分布的基因中,853个基因是亚种或品种组特异性的,包括分别为籼粳亚种,Aus和Aro组的587,147,67和52个基因。
- 水稻泛基因组的全基因组表达谱,包括226个公开可用的泛基因组表达谱的RNA-seq结果。
运用
说了那么多基本的东西,究竟这个泛基因能给作物的育种带来什么呢?下面举例子说明:
例子一:
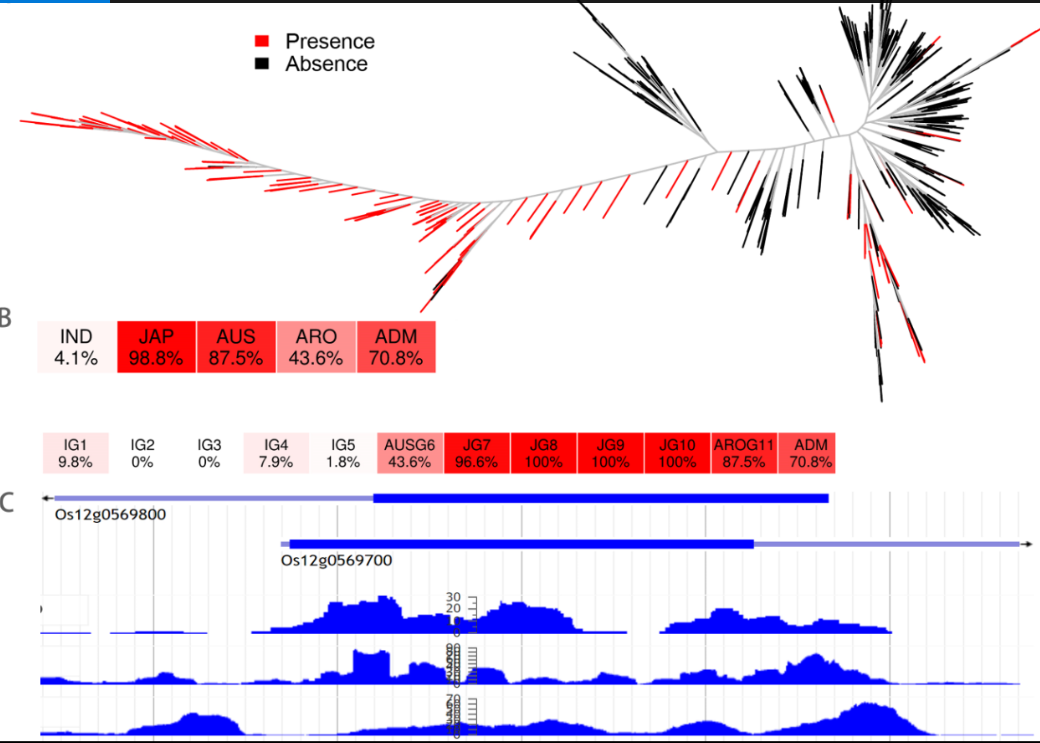
一种情况是研究人员经常搜索候选基因以获得非生物胁迫耐受性。 Os12g0569700是一个在水稻适应盐胁迫和干旱胁迫中具有潜在重要作用的基因。用户可以将基因ID Os12g0569700输入到RPAN中,并且RPAN将显示该基因存在于1107个种质中,其中795个是Japonica。该基因的频率分布和热图的系统发生树显示该基因在其他品种组中是非常低频率的粳稻显性。进一步筛选供体耐盐/耐旱性的重点应放在全长(存在)该基因的种质上。
例子二:
从3010份材料中挑选亲本用于开发早熟粳稻品种。在这项工作中,对日间长度不敏感的个体是需要选择出来的。育种者可以用控制日长不敏感的基因将候选材料加入候选材料中,而不是对全部3010份材料进行表型鉴定。已知基因Os08g0174500能够在长日照条件下抑制开花时间并调节水稻的株高和籽粒产量。用户可以首先搜索基因ID,RPAN显示Os08g0174500是一个分布基因,在品种亚组JG9中是粳稻显性的,具有最高的96.8%的频率。为了进一步找出可能的供体对携带Os08g0174500非功能性等位基因的日长不敏感,用户可以选择Os08g0174500区域中具有相对显着差异的多个tracks,点击'提交'按钮后,如基因组浏览器面板所示,可以发现一些登录号携带完整序列的Os08g0174500,而其他个体会具有不同的缺失情况。最后,根据他们在华北地区长日照(LD)条件下的开花时间,用户可能会发现B024可能是他们繁殖目的的理想供体。
总结
泛基因组的应用主要体现在提供了一个更加完善的ref genome。然后,可以从中找出更多的PAV,CNV,SNPs等变异,再将这些信息联系到表现的数据中,进而促进作物的育种。该文章中建立一个高效,可视化的基因浏览器是一个最直接有效的方法去让public得到泛基因组中的信息。