@twoer2
2019-11-30T14:59:35.000000Z
字数 1144
阅读 406
IT-vis
未分类
对于一个的网络,它的U-Matrix的形状应该是的,因为
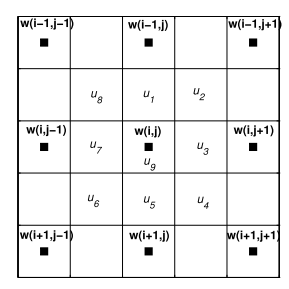
(在上图中,w代表一个neuron的权重。w(i, j)表示网络第i行第j列的neuron)
在每两个临近neuron之间,有一个数u,来衡量它们的“相似性”。如果它们之间差异太大,u就会很大,在热力图上看起来颜色就深,就像一道边界线一样把它们隔开了。
传统的SOM U-Matrix方法,使用权重的欧氏距离来衡量两个neuron之间的差异。而论文作者提出了4种新方法来衡量两个neuron之间的差异。
(1) CIP:假设每个neuron都有一个样本集。把best matching unit (BMU, 即上课说的winner)是某个neuron的样本都添加进这个neuron的里。现在估计每个neuron的的分布,两个相邻neuron之间的差异,作者认为可以用它们的的分布的差异来表示。作者用高斯分布来估计的分布,进过一轮推导,衡量两个之间的差异的公式就是CIP。
(2) rCIP: 在原始的CIP公式中,需要对两个中的每一对样本计算一次,计算量比较大。因此,作者用每个的均值和方差代表它们,将均值方差代进原来计算每对样本的公式里(2598页的G),只计算一次就够了。这个方法就是rCIP。
(3) H*: 由于CIP的值范围很大,在压缩为灰度值(0-255)的时候,可能会损失很多比较细微的差别。所以对CIP的值求log,缩小值范围,能更好地展现图片。
(4) rH*: rCIP的log版。
知道如何衡量neuron差异以后,有一个问题就是在比较小时,对分布的估计会不准。因此,作者想保证的大小不小于一个数,即MinPts。他建议两种方法:
(1) standard K-NN:将每个样本添加到它的BMU的里。添加完以后,那些大小不足MinPts的neuron,会扔掉里的所有样本,然后选MinPts个离它最近的样本添加进里(因此一个样本可能在多个里)。
(2) modified K-NN: 不会扔掉里已有的样本。每次选一个离最近的样本(样本离这个集合的距离定义为,样本里里所有的点的距离的最小值),将它添加进里。重复此步骤,直到的大小等于MinPts为止。
